Nature子刊:王杰/陈鹏合作开发“生死选择”蛋白酶进化系统,用于蛋白质的靶向编辑
来源:生物世界 2025-11-05 13:31
该研究将蛋白酶的进化与宿主的增殖实现关联,构建了一种基于“生死选择”的高通量定向进化系统。并进一步通过该系统进化 TEV 蛋白酶,使其能识别并切割多种细胞靶蛋白。
蛋白酶通过切割酶原或前体蛋白生成新的生物活性分子,参与调控多种生理与病理过程,包括程序性细胞死亡、血液凝固级联反应以及病原体感染等。重编程蛋白酶底物特异性,有望实现对目标蛋白的选择性降解、特定信号通路的按需激活,甚至病理性蛋白的精准清除,从而衍生一系列在基础研究与疾病干预中的革命性应用。
对标基因编辑工具对核酸序列进行靶向切割(或改写),若能以类似的“可编程”理念重塑蛋白酶的底物特异性,使其识别并切割新的目标蛋白,将为蛋白质层面的精准编辑开辟全新路径。基因编辑需要识别 15-18 个碱基长度的核酸序列才可以在基因组上实现特异性不同,蛋白酶仅需要识别 5-7 个氨基酸长度的多肽序列就可以在蛋白组上实现特异性,而一个蛋白酶的口袋大小,通常在 4-8 个氨基酸范围,因此,通过高效的定向进化方法,改变蛋白酶的底物特异性,有望实现蛋白质的靶向编辑。
2025 年 10 月 31 日,南方科技大学王杰团队与北京大学陈鹏团队合作(南方科技大学博士后高子奇、李天真和研究生叶昊为论文共同第一作者)在 Nature 子刊 Nature Chemical Biology 上发表了题为:A gasdermin-based life–death evolutionsystem for reprogramming protease specificity 的研究论文。
该研究将蛋白酶的进化与宿主的增殖实现关联,构建了一种基于“生死选择”的高通量定向进化系统。并进一步通过该系统进化 TEV 蛋白酶,使其能识别并切割多种细胞靶蛋白。

天然蛋白酶的序列多样性有限,难以满足大规模、精确的蛋白质编辑需求。要实现这一目标,必须对天然酶的底物特异性进行系统性重编程。然而,要让一种蛋白酶“改刀换刃”并非易事。蛋白酶识别底物的方式极为精细:它们依赖 4–8 个连续氨基酸残基在空间和化学上的精确匹配,以定位切割位点。哪怕微小的识别序列改变,也可能扰动酶的整体构象或催化中心,导致活性丧失。传统的定向进化方法虽然可以通过突变和筛选获得部分改良的酶,但受限于通量和成本,难以在庞大的突变空间中快速筛选出理想变体,实现对不同序列的高特异性切割。这使得蛋白酶的特异性改造长期停留在“微调”阶段,而难以实现真正意义上的重编程。
为了实现对蛋白酶底物特异性的重塑,研究团队利用细胞焦亡的功能执行蛋白-Gasdermin D(GSDMD),构建了一种基于“生死选择”策略的蛋白酶定向进化平台。
首先,他们发现,在原核生物中表达 GSDMD 的 N 端结构域(1-275,GD-N)可以对大肠杆菌的膜结构产生破坏,从而对其生长产生明显的抑制。不仅如此,大肠杆菌的生长速度可以很好的受到 GD-N 的表达水平的调控,呈现出剂量依赖的效果,从而验证 GD-N 是一个很好的筛选标签。
随后,他们在 GD-N 蛋白表面的 loop 区(R153-V163)插入了TEV蛋白酶的目标序列,通过筛选发现这一序列的引入不影响 GD-N 蛋白对大肠杆菌生长的抑制,进而实现对蛋白酶突变体的筛选:当蛋白酶突变体能识别并切割目标序列,毒蛋白 GD-N 的活性被破坏,细胞存活;当蛋白酶突变体不能识别目标序列,GD-N 蛋白对细菌膜结构破坏,细菌生长受到抑制。
通过连续的培养,含有高活性突变体的大肠杆菌,生长速度较快,从而被富集,构建好的蛋白酶定向进化平台可一轮筛选中,对高活性突变体可以实现百万倍的富集效果。
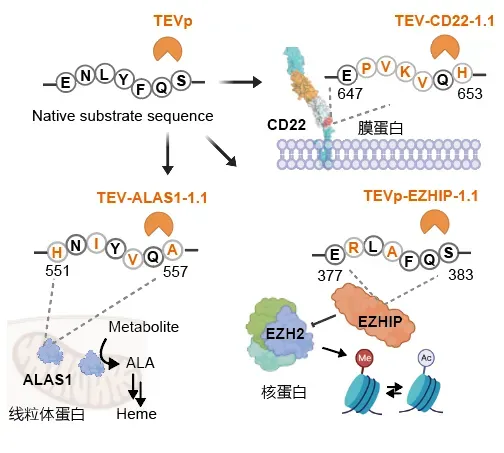
研究团队利用所建立的定向进化平台,对 TEV 蛋白酶进行了系统进化,使其能够特异性识别并切割来源多样、结构复杂的内源性底物,包括膜蛋白 CD22、线粒体蛋白 ALAS1 以及核蛋白 EZHIP。其中 CD22 是一种 B 细胞特异性膜受体,通过抑制 BCR 信号维持免疫稳态,同时是 B 细胞相关肿瘤的重要治疗靶点。ALAS1 是血红素生物合成途径中的关键限速酶,其异常激活是 Ⅰ 型卟啉病的主要致病因素。EZHIP 是一种位于细胞核的调控蛋白,通过抑制 PRC2 活性重塑 H3K27 甲基化图谱,在维持发育可塑性及中枢神经系统肿瘤发生中发挥关键作用;其异常高表达可导致表观遗传失衡并促发弥漫性中线胶质瘤。
对于 CD22 靶点,研究团队选取了其靠近细胞膜结构域的一段序列,该序列与 TEV 原始识别序列相比,在 7 个氨基酸中有 5 处发生了改变(ENLYFQS vs. EPVKVQH)。基于对底物不同氨基酸位点对特异性贡献的分析,以及识别口袋中关键残基在底物结合中的作用,团队设计了 TEV 蛋白酶的进化路径,并构建了满足多轮进化需求的饱和点突变文库,最终获得了能够高效切割目标序列的酶切活性突变体。
对于 ALAS1 靶点,研究团队选取了 HNIYVQA(551-557)作为目标序列, 研究团队通过定向进化获得了能够特异性识别并切割 ALAS1 特定氨基酸序列的 TEV 蛋白酶突变体。在体外和体内实验中,该突变体均表现出良好的序列正交性,未观察到明显脱靶效应。
对于 EZHIP 靶点,研究团队选取了 ERLAFQS(377-383)作为目标序列,研究团利用 GSDMD 进化平台,仅一轮进化便获得了能够特异性酶切 EZHIP 的 TEV 突变体。该突变体在体外对 EZHIP 表现出明显的切割活性;将其递送至 U2OS 细胞后,能有效降低内源性 EZHIP 蛋白水平,同时显著提高组蛋白 H3K27Me3 水平。
蛋白酶的特异性进化与蛋白质的靶向编辑
总的来说,这项工作建立的 GSDMD 定向进化平台,能够高效重编程蛋白酶底物特异性,实现对多种内源性蛋白的精准切割。该平台可在单轮筛选中实现百万倍富集,显著提高筛选效率;结合饱和点突变库和底物-逐步策略,能够覆盖超过 10⁹ 级别的蛋白酶突变体,充分探索突变空间。利用该平台成功进化 TEV 蛋白酶切割膜蛋白 CD22、线粒体蛋白 ALAS1 及核蛋白 EZHIP,验证了其在不同亚细胞环境下的普适性与可编程性。此外,该平台可与多种蛋白递送系统结合,实现内源性蛋白在活细胞中的定点切割,为蛋白质组水平的精确编辑提供强有力工具,同时为治疗、诊断及功能研究应用奠定了基础。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


